| 题目名称 | 1916. [CF 520C]DNA Alignment |
|---|---|
| 输入输出 | cf520C.in/out |
| 难度等级 | ★☆ |
| 时间限制 | 1000 ms (1 s) |
| 内存限制 | 256 MiB |
| 测试数据 | 25 |
| 题目来源 |
|
| 开放分组 | 全部用户 |
| 提交状态 | |
| 分类标签 | |
| 分享题解 |
| 通过:3, 提交:4, 通过率:75% | ||||
|
|
100 | 0.014 s | 0.41 MiB | C++ |
|
|
100 | 0.015 s | 0.29 MiB | C++ |
|
|
100 | 0.016 s | 0.41 MiB | C++ |
|
|
48 | 0.016 s | 0.39 MiB | C++ |
| 关于 DNA Alignment 的近10条评论(全部评论) | ||||
|---|---|---|---|---|
|
=_=第一次补cf题好紧张啊……
| ||||
【题目描述】
对于两个长度均为N的字符串s, t,定义:
$h(s, t)$为两个字符串中字符相同的位置个数,
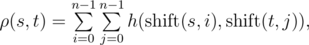
其中$shift(s, i)$表示将字符串s循环左移得到的字符串。
例如:ρ("AGC", "CGT") = h("AGC", "CGT") + h("AGC", "GTC") + h("AGC", "TCG") + h("GCA", "CGT") + h("GCA", "GTC") + h("GCA", "TCG") + h("CAG", "CGT") + h("CAG", "GTC") + h("CAG", "TCG") = 1 + 1 + 0 + 0 + 1 + 1 + 1 + 0 + 1 = 6.
现在,你有一个长度为N 的DNA串S。你希望求出所有使得$\rho(S, T)$取最大值的DNA串T 的个数。换句话说,你要求出满足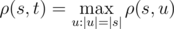 的长度为N 的DNA串T 的个数。其中DNA串表示所有字符均为A,G,C,T的字符串。
的长度为N 的DNA串T 的个数。其中DNA串表示所有字符均为A,G,C,T的字符串。
由于这个答案可能很大,你只需输出这个数除以$10^9 + 7$的余数。
【输入格式】
第一行是一个正整数N,表示DNA串的长度。
第二行是一个长度为N 的DNA串S。
【输出格式】
一行一个整数。
【样例输入1】
1 C
【样例输出1】
1
【样例输入2】
2 CG
【样例输出2】
4
【样例输入3】
3 TTT
【样例输出3】
1
【数据范围与提示】
$1 \leq N \leq 10^5$
字符串中只包含A,G,C,T.
NOTE:
Please note that if for two distinct strings t1 and t2 values ρ(s, t1) и ρ(s, t2) are maximum among all possible t,
then both strings must be taken into account in the answer even if one
of them can be obtained by a circular shift of another one.
In the first sample, there is ρ("C", "C") = 1, for the remaining strings t of length 1 the value of ρ(s, t) is 0.
In the second sample, ρ("AG", "AG") = ρ("AG", "GA") = ρ("AG", "AA") = ρ("AG", "GG") = 4.
In the third sample, ρ("TTT", "TTT") = 27
【来源】
CF #295 div1 A